kaggle UBC OC 대회 기록 01
업데이트:
kaggle Ovarian Cancer Subtype Classification and Outlier Detection

1일차 기록
kaggle에서 진행하는 Ovarian Cancer Subtype Classification and Outlier Detection (난소암 서브타입 분류 / 이상치 탐지) 대회를 참여한지는 시간이 꽤 지났지만, 수업과 논문스터디를 병행하며 시간이 없었기 때문에 이제서야 간단한 EDA와 전처리(resize)를 진행하였다.
대회에서 train/test_data의 용량만 총 718GB이기 때문에, kaggle에서 dataset을 다운받는데만 약 18시간이 걸렸고, train_image는 한 장이 GB단위 크기의 png이미지이기 때문에 train_data folder에 접근하기만 해도 탐색기가 응답불가가 떠버린다…
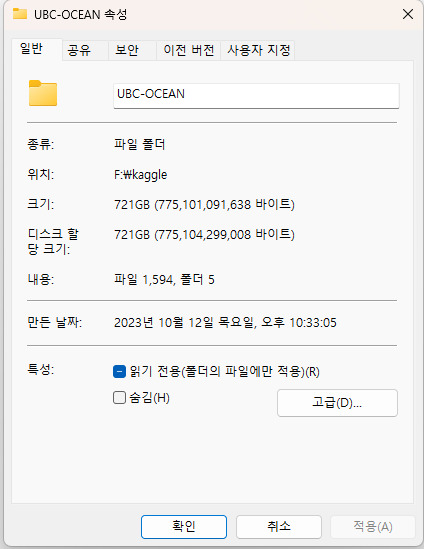
EDA?
이미지는 전체 슬라이드 이미지(WSI - 20배율)와 조직 마이크로 어레이(TMA - 40배율)의 두 가지 종류가 있다.
가장 큰 이미지는 100,000 x 50,000 pixel에 달하는 어마어마한 크기를 자랑하며, 이미지의 크기와 품질, 슬라이드 염색 기법 등의 차이 등을 포함해서 여러 방면으로 꼼꼼히 이미지를 확인하라는 조언도 달려있다.
test_data는 약 2,000장의 이미지가 포함되며, 대부분이 TMA이미지이고, 550GB여서 kaggle notebook에서 load하는데 굉장히 오래 걸리며, 모델이 얼마나 잘 일반화 되었는지를 판단하기 위해 특별히 구성되었다고 한다.
우리가 분류해야할 난소암의 하위유형은
- CC
- EC
- HGSC
- LGSC
- MC
- 기타
로 이루어져 있다. (기타 클래스는 train_data에 존재하지 않으므로 이상값으로 생각하고 식별해 내는것도 task의 일부)
또한 해당 이미지가 TMA인지 아닌지를 판별해주는 is_tma 라벨이 존재하며, TMA는 train_thumbnail이 제공되지 않는다.
WSI? TMA?
WSI
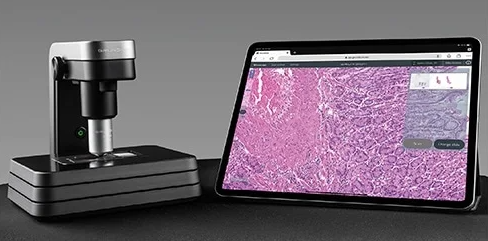
- Whole Slide Imaging
- 조직 샘플 또는 조직 슬라이드의 전체 이미지를 고해상도로 디지털화하는 기술.
TMA
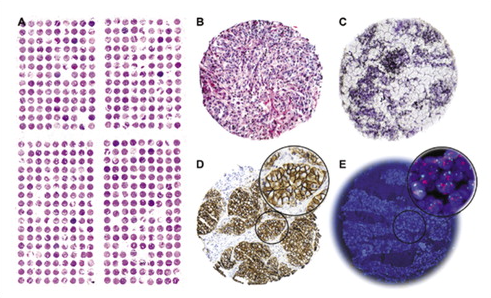
- Tissue MicroArray
- 여러개의 조직 샘플을 한 슬라이드에 배열하여 효율적으로 조직 분석 및 비교를 할 수 있게 해주는 기술.
- 주로 종양학 연구나 조직 분석에서 사용됨.
Image Resizing
원본 이미지의 크기가 어마어마하게 크기 때문에, opencv로 이미지를 읽어온 후 resize를 통해 1024 x 1024 pixel로 resizing을 진행했다.
resizing을 진행하기 위해 cv2로 이미지를 읽어오는데, 이미지가 너무 커서 읽어올 수 없었다🤔
먼저 cv2의 image size 제한을 풀어주었다.
import os
os.environ["OPENCV_IO_MAX_IMAGE_PIXELS"] = pow(2,40).__str__()
import cv2 # import after setting OPENCV_IO_MAX_IMAGE_PIXELS
위에 쓰여있는대로, environ 설정을 먼저 해준 뒤에 cv2를 import해야 적용된다!
그 다음으로 큰 이미지를 작은 이미지로 축소할 때 보간법을 선택해야 하는데, cv2에 있는 5가지 보간법을 비교해보며 가장 좋은 보간법을 찾아보았다.
interpolation_list = [cv2.INTER_NEAREST, cv2.INTER_LINEAR, cv2.INTER_CUBIC, cv2.INTER_LANCZOS4, cv2.INTER_AREA]
interpolation_slist = ['INTER_NEAREST', 'INTER_LINEAR', 'INTER_CUBIC', 'INTER_LANCZOS4', 'INTER_AREA']
for i in zip(interpolation_list, interpolation_slist):
resized_image = cv2.resize(image, (1024, 1024), interpolation=i[0])
# resized_image.shape
cv2.imwrite(f'{resized_train_image_dir}{i[1]}.jpg', resized_image)
일반적으로 cv2.INTER_AREA가 이미지를 축소할 때 가장 성능이 좋다고 했는데, 테스트 결과 역시나 가장 부드러운 결과물이 나오게 되어 cv2.INTER_AREA로 결정하게 되었다. (각 보간법이 적용된 image를 올리고 싶지만, kaggle이 train data의 embago가 풀리기 전에는 외부로 데이터를 유출하면 안된다고 했기 때문에 나중에 embago가 풀리면 올려보도록 하겠다🫡)
마지막으로 interpolation=cv2.INTER_AREA를 적용한 후 1024 x 1024 size로 resize된 image를 .jpg 확장자로 변환해 따로 저장해 주었다.
for img in tqdm.tqdm(train_image_list):
image = cv2.imread(train_image_dir + img)
resized_image = cv2.resize(image, (1024, 1024), interpolation=cv2.INTER_AREA)
cv2.imwrite(f'{resized_train_image_dir}{img[:-4]}.jpg', resized_image)
del image
(del image는 혹시모를 memory issue를 방지하기 위해 넣어주었다.)
그 결과..
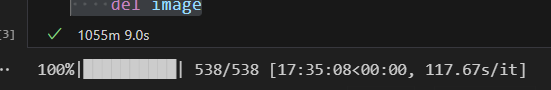
17시간 35분 8초가 걸려서 모든 이미지의 resizing이 끝났다!
TO-DO?
이렇게 모든 이미지를 resizing 하였는데, 결과물을 확인하던 중 문제점을 발견했다.
모든 이미지가 W, H 비율이 비슷한게 아닌, 두 개 이상의 tissue sample이 붙어있는 가로로 긴 이미지들도 꽤 존재한다는 것! (마찬가지로 embago가 풀려야 이미지를 올릴 수 있다 ㅠㅠ)
내일은 이 가로로 긴 이미지를 어떻게 처리해야 할지 고민을 좀 더 해봐야 할 것 같다.

댓글남기기